Geovanni Ramos Velasco
Químico farmacobiólogo. Unidad de Especialidades Médicas-Enfermedades Crónicas. Jurisdicción Sanitaria II. Secretaría de Salud. San Cristóbal de Las Casas (Chiapas, México)
Epigenética. Bases moleculares. Factores ambientales. Cambios epigenéticos. Diabetes mellitus tipo 2.
DOI: 10.26322/2013.7923.20170519.3
La diabetes mellitus tipo 2 es una enfermedad crónica multifactorial, caracterizada por hiperglucemia. Los elevados niveles de glucosa en sangre pueden dañar órganos y llevar a complicaciones del sistema cardiovascular, ojos, neuronas y riñones, entre otros. Actualmente es una de las enfermedades más frecuentes, y su incidencia está aumentando a una velocidad impresionante en todo el mundo. Recientes estudios de asociación han identificado una serie de factores genéticos que explican parte de la variación interindividual en la predisposición a la diabetes mellitus tipo 2. También hay un creciente cuerpo de literatura que sugiere un papel para los factores epigenéticos en la compleja interacción entre los genes y el medio ambiente. La epigenética se define como los cambios en la función génica que no son codificados por el genoma; no se dan cambios estructurales en los genes, no hay mutaciones, lo que habrá será una especie de cambios químicos reversibles. Los factores epigenéticos incluyen metilaciones del ADN, modificaciones de las histonas y microácido ribonucleico (micro-ARN). Un ejemplo de estos mecanismos se da en el gen INS (responsable de producir insulina); al estar metilado, se suprime la expresión y por consiguiente, la insulina no se producirá. Es evidente que los mecanismos epigenéticos desempeñan un papel importante en la patogénesis de la diabetes mellitus tipo 2 y sus complicaciones. El uso de tecnologías de todo el genoma para estudiar los cambios epigenéticos ayudará a identificar genes candidatos que pueden ser dianas terapéuticas, y también permitirá diseccionar en profundidad el papel de la metilación del ADN y las modificaciones a las histonas en el desarrollo de la diabetes mellitus tipo 2.
Este artículo de revisión pretende analizar el papel de los cambios epigenéticos que se presentan en la diabetes mellitus tipo 2 y evidenciar que los factores ambientales y genéticos son un desencadenante importante de la enfermedad y sus complicaciones.
1. Contexto
La diabetes mellitus tipo 2 (DM2) es una enfermedad crónica multifactorial caracterizada por hiperglucemia, un resultado del deterioro en la función de las células β pancreáticas y resistencia a la insulina en tejidos como el músculo esquelético y el tejido adiposo. En la resistencia a la insulina, las células corporales muestran una respuesta reducida a la insulina, lo que a su vez aumenta la glucosa en la sangre, y esto obliga a las células β a compensar produciendo más insulina. Al final la pérdida de equilibrio deriva en hiperglucemia. Los elevados niveles de glucosa en sangre (y deficiencia en los tejidos) pueden dañar órganos y llevar a complicaciones del sistema cardiovascular, ojos, neuronas y riñones, entre otros1.
La DM2 es, actualmente, una de las enfermedades más frecuentes, y su incidencia está aumentando a una velocidad impresionante. Constituye una de las principales causas de morbimortalidad del mundo. Este aumento de la tasa puede explicarse, en parte, por la mejora en los métodos diagnósticos y por un tratamiento más efectivo de la enfermedad, lo que ha incrementado la esperanza de vida del paciente con diabetes mellitus (DM). De acuerdo con la International Diabetes Federation, la prevalencia de la DM se estimó en 35 millones de pacientes con DM en América; de ellos, 19 millones (54 %) residían en América Latina y el Caribe. En el mundo occidental se estima una prevalencia que oscila entre el 2 y el 6 % de la población, según los diferentes estudios, con el agravante de que más del 50 % de los casos permanecen sin diagnosticar; es decir, por cada persona con DM conocida existe una con DM desconocida2.
Aunque la obesidad, la reducción de la actividad física y el envejecimiento aumentan la propensión a la DM2, muchas personas expuestas a estos factores de riesgo no desarrollan la enfermedad. Recientes estudios de asociación han identificado una serie de factores genéticos que explican parte de la variación interindividual en la predisposición a la DM. También hay un creciente cuerpo de literatura que sugiere un papel para los factores epigenéticos en la compleja interacción entre los genes y el medio ambiente. Podríamos definir la epigenética como el proceso de cambiar la función de un gen sin un cambio en la secuencia de nucleótidos. En particular, los efectos epigenéticos también pueden verse afectados por el medio ambiente, haciéndolos mecanismos patógenos potencialmente importantes en enfermedades multifactoriales complejas, como la DM23.
2. ¿Qué es la epigenética?
Son los cambios en la función génica que no son codificados por el genoma (son independientes de la secuencia de ácido desoxirribonucleico [ADN]). Son procesos que suceden por encima de la genética, en relación con la cromatina. Hace referencia, en un sentido amplio, al estudio de todos aquellos factores no genéticos que intervienen en la determinación de la ontogenia. Es la regulación heredable de la expresión genética sin cambios en la secuencia de nucleótidos4.
No se dan cambios estructurales en los genes, no hay mutaciones en la secuencia del genoma; lo que habrá será una especie de cambios químicos reversibles (cambios estocásticos) que dan lugar a cambios en la expresión de los genes. Por ejemplo, se dan cambios químicos como la metilación. Al analizar a dos gemelas homocigóticas, en principio parten con el mismo genoma, pero una de ellas desarrolla esquizofrenia: los sitios de metilación son similares entre los gemelos al inicio de sus vidas, pero conforme van pasando los años estos sitios de metilación van cambiando y van siendo cada vez más distintos entre ellos4.
Grandes estudios epidemiológicos nos han permitido ver la gran importancia de la regulación epigenética en el condicionamiento del desarrollo embrionario o adaptabilidad de los embriones. Esto se dio al final de la segunda guerra mundial en Amsterdam, donde se bloqueó el acceso a los alimentos. Las madres gestantes dieron lugar a niños pequeñitos. Los nietos de estos niños desarrollaron enfermedades como DM. Se encontró que el gen de la insulina tenía un patrón aberrante de metilación, porque eran niños preparados para dietas poco calóricas4.
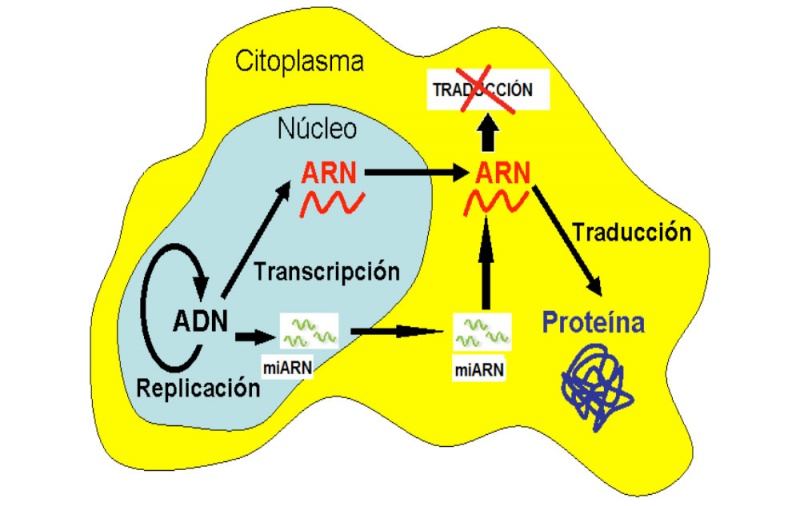
A diferencia de nuestro genoma, el epigenoma es muy dinámico y va cambiando con el tiempo (figura 1).
Otra manera de explicar el epigenoma son las modificaciones químicas en unas proteínas llamadas histonas que empaquetan nuestro ADN en nucleosomas, que son capaces de abrir o cerrar nuestra cromatina y hacer que nuestros genes se expresen o se silencien4.
2.1 Bases moleculares
Los factores epigenéticos incluyen metilaciones del ADN, modificaciones de las histonas y microácido ribonucleico (micro-ARN). Estos mecanismos pueden ayudar a explicar cómo las células con ADN idéntico pueden diferenciarse en distintos tipos de células con diferentes fenotipos3.
2.2 Metilación del ácido desoxirribonucleico
La metilación del ADN (figura 2) es la principal modificación epigenética del genoma y regula aspectos fundamentales de su función. Es un mecanismo de silenciamiento génico con un evidente papel en la represión transcripcional. La metilación del ADN no ocurre en cualquier sitio, sino que existen dianas específicas del genoma donde se cataliza esta reacción. En los seres humanos el ADN se metila únicamente en las citosinas, y más específicamente, en citosinas que estén unidas a guaninas a través de un enlace fosfato, es decir, en dinucleótidos «citosina-fosfato-guanina» o CpG. La adición del grupo metilo a la citosina da lugar al nucleótido 5-metil-citosina, reacción que es catalizada por un grupo de enzimas conocidas como ADN metiltransferasas (DNMT). La mayor parte del genoma no contiene el dinucleótido CpG; este se concentra en las denominadas «islas CpG», situadas en las regiones reguladoras de los genes, posicionadas en el extremo 5’ de muchos genes5.
La reacción de metilación del ADN es catalizada por las DNMT e involucra la transferencia del grupo metilo de la S-adenosil-L-metionina al carbono 5 de la citosina. En las células de los mamíferos se han identificado tres enzimas diferentes que llevan a cabo esta reacción: las DNMT de «mantenimiento» (DNMT1) y las metilasas de novo (DNMT3A y DNMT3B)6.
2.3 Modificación postraduccional de histonas
El nucleosoma (figura 3) es la unidad fundamental de la cromatina. Las histonas que forman la médula de esta unidad funcional son pequeñas y altamente básicas. Están compuestas por un dominio globular y un dominio N-terminal, flexible y poco estructurado, conocido como «cola de histonas», que protruyen fuera del nucleosoma. Ambos dominios son dianas para modificaciones químicas. El efecto de estas modificaciones altera la naturaleza del nucleosoma de tal forma que «abre» o «cierra» a la cromatina para realizar la transcripción.
ADN: ácido desoxirribonucleico
Diferentes combinaciones específicas de modificaciones covalentes y no covalentes de las histonas pueden leerse como un código, lo cual se conoce como hipótesis del «código de histonas». Estas modificaciones son reguladas por grupos de diferentes enzimas que pueden añadir grupos químicos a las histonas (marcadores o writers) o eliminar grupos químicos (borradores o erasers). Se han encontrado al menos ocho tipos de modificaciones postraduccionales en las histonas. Las «colas» de las histonas pueden sufrir acetilación, metilación, fosforilación, poli-ADP (difosfato de adenosina)-ribosilación, ubiquitinación y glucosilación. Las modificaciones de las histonas (figura 4) funcionan mediante dos mecanismos básicos:
Una de las modificaciones de histonas mejor estudiadas es la acetilación. Esta reacción es catalizada por «histonas acetiltransferasas». La acetilación de histonas ocurre, principalmente, en los residuos de lisina de las histonas H3 y H4 y tiene, básicamente, dos consecuencias biológicas: alteración de la unión «histona-ADN», ya que la lisina pierde una carga positiva en el proceso, y alteraciones en los códigos de acoplamiento de los factores de transcripción que interactúan con la cromatina. Así, la acetilación posee un efecto activador de la transcripción; los nucleosomas se empaquetan menos eficientemente, lo que permite que el ADN sea más accesible a proteínas reguladoras. Otro mecanismo bien conocido es la metilación de histonas. Se vincula tanto a activación como a represión transcripcional.
Las «colas» de las histonas pueden ser metiladas en múltiples residuos de arginina y lisina. La metilación es catalizada por una familia de enzimas conocidas como «histonas metiltransferasas»8.
ADN: ácido desoxirribonucleico; C: citosina; G: guanina; p: enlace de fosfato.
2.4 Ácido ribonucleico no codificante
Uno de los avances más importantes en la biología de las últimas décadas ha sido el descubrimiento de moléculas de ácido ribonucleico (ARN) que regulan la expresión de genes. El silenciamiento por ARN (figura 5) es un mecanismo altamente conservado en la naturaleza, en el que moléculas de ARN regulan la expresión de genes. Se han descrito varias clases de moléculas pequeñas de ARN que desencadenan el proceso de silenciamiento por interferencia. Las más ampliamente estudiadas son los ARN interferentes pequeños (si-ARN) y los micro-ARN. Los micro-ARN son pequeños ARN (18-25 nucleótidos) endógenos, no codificadores de proteínas, que impiden la expresión de un determinado gen. Esto lo logran bloqueando la traducción (mecanismo antisentido) o mediando la degradación de ARN mensajeros específicos (aquellos poseedores de una secuencia complementaria al micro-ARN). Fisiológicamente, los micro-ARN ejercen varias funciones. Desde el punto de vista epigenético, no solo tienen la capacidad de regular la expresión génica mediante los mecanismos expuestos anteriormente, sino que también pueden remodelar la cromatina al modificar el patrón de metilación de una secuencia específica. También se han visto implicados en la corrección selectiva de errores en la metilación del ADN, lo que protegería al organismo contra una eventual pérdida transgeneracional del patrón de metilación9.
ADN: ácido desoxirribonucleico; ARN: ácido ribonucleico; mi-ARN: microácido ribonucleico.
Los mecanismos epigenéticos están íntimamente interrelacionados; todos ellos trabajan a niveles distintos de la organización genética, pero todos dan como resultado un conjunto de modificaciones relevantes de la estructura cromatínica. Así pues, la modificación de una histona puede llevar a la metilación del ADN; esta, a su vez, contribuye a una mayor compactación del ADN sobre las histonas, y se crea así una cromatina más cerrada. Esta región será menos accesible a la maquinaria de transcripción, por lo que el gen que regulan se silencia y no se codifica su producto. De manera ilustrativa, comparamos los sistemas epigenéticos con un sencillo sistema de «encendido/apagado». Cuando el gen está «apagado», metilado, reprimido, no se transcribirá su información genética. Es interesante considerar que este proceso puede ser reversible, por lo que un gen que se encuentra apagado puede activarse nuevamente.
3. Epigenética y diabetes mellitus tipo 2
En 1992 se propuso que los factores ambientales experimentados en la vida temprana podían aumentar el riesgo de DM2 en la vida posterior. En particular, la desnutrición y el bajo peso al nacer mostraron una relación con la DM2, la resistencia a la insulina y el deterioro en la secreción de insulina en la vida adulta. La nutrición inadecuada, al inducir alteraciones crónicas en el metabolismo, en los niveles de hormonas y en el número de células, contribuye al riesgo de DM2. La plasticidad en el desarrollo hace posible para el embrión humano temprano adaptarse a su ambiente en un momento dado, pero cuando la situación ambiental cambia, después en la vida, el beneficio del mejor uso de los nutrimentos se vuelve una desventaja10.
Las células β pancreáticas sintetizan y secretan insulina. La regulación de la expresión del gen de insulina (INS) no se comprende del todo, pero existe evidencia de involucramiento epigenético, tanto de estudios sobre la estructura de la cromatina como en el nivel de metilación del ADN.
Las células madre embrionarias tienen un patrón intermedio de marcas epigenéticas, consecuente con su potencial para diferenciarse en una célula que expresa insulina. En estas células, el gen INS despliega un patrón de cromatina típico de los genes activos, los sitios CpG de los promotores (región del ADN que controla la iniciación de la transcripción de un gen) humanos INS están desmetilados y, por lo tanto, la maquinaria de transcripción puede iniciar el proceso que llevará a la formación de la insulina, ya que la cromatina estará abierta, pero la metilación de estos sitios suprime la expresión génica (cromatina cerrada); por consiguiente, la insulina no se producirá10.
Por otro lado, los pacientes con DM, como resultado de la acumulación de glucosa, incrementan la glicación de varias proteínas, entre ellas la hemoglobina glucosilada (HbA1c). La HbA1c se forma cuando la glucosa se une a la hemoglobina A. La formación de HbA1c es irreversible, y su concentración en sangre depende de la vida media de los hematíes (alrededor de 120 días) y de la concentración de glucosa en sangre. Así, la medición de la HbA1c es una prueba de laboratorio muy utilizada como criterio diagnóstico de DM (valores de HbA1c ≥ 6,5 %) y para el control de los niveles de glucosa en sangre de pacientes con DM en los últimos dos meses.
Como ya se mencionó, la DM es una enfermedad compleja en la que tanto factores genéticos como ambientales contribuyen a su desarrollo. Los mecanismos epigenéticos, como la metilación del ADN, se ha asociado a procesos de envejecimiento y a la modulación del riesgo de sufrir varias patologías, como la DM. La metilación del ADN podría ser un mecanismo mediador de la relación de DM con exposiciones ambientales o hábitos de vida, como la dieta11.
Un estudio realizado por el Departamento de Neurología del Hospital del Mar, en conjunto con el Grupo de Investigación en Epidemiología y Genética Cardiovascular del Instituto Hospital del Mar de Investigaciones Médicas y la Universitat Pompeu Fabra, identificó cambios de metilación asociados a la DM. Para ello llevaron a cabo un experimento de metilación del genoma a partir de ADN de sangre periférica de una cohorte de pacientes con infarto cerebral del Hospital del Mar de Barcelona. Usando el chip de Illumina, HumanMethylation450 BeadChip®, que permite estudiar el estado de metilación de más de 480 000 posiciones de metilación del genoma humano. Todos los análisis estadísticos se ajustaron según las variables sexo, edad, hiperlipidemia, índice de masa corporal, hábito de fumar y recuento celular sanguíneo. El hallazgo se replicó en dos cohortes independientes, otra cohorte de pacientes con ictus (165 pacientes) y otra cohorte de origen poblacional (645 sujetos).
En la fase de descubrimiento, donde se analizaron 355 pacientes (de los cuales 151 presentaban DM2), se identificó un punto de metilación CpG 19693031 localizado en el gen TXNIP, hipometilado en el grupo de pacientes con DM. Este hallazgo se replicó en las dos cohortes independientes. Además, la metilación de TXNIP se asociaba de forma inversamente proporcional a los niveles de HbA1c, es decir, a mayor porcentaje de HbA1c, menor metilación en CpG 19693031 de TXNIP.
La asociación es específica de pacientes con DM, pero concretamente en aquellos que presentaban un mal control de sus niveles de glucosa en los últimos dos meses, indicado por valores iguales o superiores al 7 % de HbA1c.
El cambio de metilación de TXNIP podría utilizarse como biomarcador temprano de disfunción del control de los niveles de glucosa y en un futuro podría ser una posible diana terapéutica en el tratamiento de la DM o en el control de la concentración de glucosa11.
Aunque existe apoyo para el papel de la epigenética en la patogénesis de la DM2, los estudios concluyentes en tejidos humanos son limitados. En un estudio, la S-adenosilmetionina, el principal donador fisiológico de grupos metilo, estuvo disminuido en los eritrocitos de pacientes con DM2. Asimismo, una disminución en el donador de metilo estuvo asociada con la progresión de la enfermedad. En efecto, el tratamiento con S-adenosilmetionina mejora la sensibilidad a la insulina en un modelo de ratón con resistencia a la insulina y DM2, debido, posiblemente, a un incremento en la densidad de ADN mitocondrial en el músculo esquelético. El siguiente paso es probar este tratamiento en tejidos humanos10.
Un estudio funcional que evaluó la epigenética en el tejido humano con DM2 concierne al coactivador 1-α (PGC-1α), codificado por el gen PPARGC1A. Se trata de un coactivador transcripcional de genes mitocondriales involucrado en la producción normal de ATP (trifosfato de adenosina) y la secreción de insulina por las células β pancreáticas.
El estudio mostró que el nivel de metilación de ADN se incrementa en una región promotora (iniciadora de transcripción) de PPARGC1A en las isletas pancreáticas de pacientes con DM2, cuando se comparan con las isletas de donantes humanos sanos.
Este aumento en la metilación de ADN se correlaciona con una disminución en la expresión del ARN mensajero de PPARGC1A; asimismo, la expresión de PPARGC1A está positivamente correlacionada con la secreción de insulina estimulada por glucosa. Más aún, en el músculo esquelético de pacientes con DM2, un incremento en la metilación del ADN es paralelo a un decremento en la expresión de ARN mensajero de PPARGC1A y al contenido mitocondrial; por lo tanto, al estar metilada la región promotora de este gen, la producción de insulina se verá limitada o será casi nula, con la consiguiente elevación de glucosa en sangre10.
4. Conclusiones
Como en otras patologías, la DM2 es el resultado de interacciones complejas, entre factores ambientales y genéticos, durante la vida de un individuo. Basándonos en el conocimiento actual, es evidente que los mecanismos epigenéticos desempeñan un papel importante en la patogénesis de la DM2 y sus complicaciones. Sin embargo, estamos tan solo comenzando a comprender cuáles son y cómo los factores epigenéticos afectan e intervienen en esta enfermedad. El uso de tecnologías de todo el genoma para estudiar los cambios epigenéticos en los tejidos objetivo de DM2, tanto en grupos de riesgo antes de que la enfermedad se haya desarrollado como en pacientes con DM2, ayudará a identificar genes candidatos que pueden ser dianas terapéuticas.
Sumado a esto, el estudio del epigenoma ayudará a desarrollar herramientas y metodologías que predigan, diagnostiquen y traten la enfermedad. Con la ayuda de más estudios del genoma completo, puede ser posible diseccionar en profundidad el papel de la metilación del ADN y las modificaciones a las histonas en el desarrollo de la DM2.
1. King H, Aubert RE, Herman WH. Global burden of diabetes, 1995-2025. Diabetes Care 1998;21:1414-31.
2. Avilán Rovira JM. Epidemiología de la diabetes en Venezuela. Gac Med Caracas 2004;112.
3. Storino-Farina MA, Contreras-Zambrano MA. Epigenética y diabetes: el rol de las DPP-4, artículo de revisión. Revista de Endocrinología y Nutrición 2012;20:169-74.
4. García Giménez JL. Regulación epigenética y su importancia clínica. Material didáctico. Módulo 2: Introducción a la genética médica, clase 7. Valencia: Universidad de València (ADEIT Fundación Universidad-Empresa de la Universidad de Valencia); 2015: p. 1-13.
5. Webster AL, Yan MS, Marsden PA. Epigenetics and cardiovascular disease. Can J Cardiol 2013;29:46-57.
6. Udali S, Guarini P, Moruzzi S, Choi SW, Friso S. Cardiovascular epigenetics: from DNA methylation to microRNAs. Mol Aspects Med 2013;34:883-901.
7. Handel AE, Ebers GC, Ramagopalan SV. Epigenetics: molecular mechanisms and implications for disease. Trends Mol Med 2010;16:7-16.
8. Lorenzen JM, Martino F, Thum T. Epigenetic modifications in cardiovascular disease. Basic Res Cardiol 2012;107:245.
9. Moore LD, Le T, Fan G. DNA methylation and its basic function. Neuropsychopharmacology 2013;38:23-38.
10. Edid M. Epigenética y diabetes mellitus tipo 2. 2011. Disponible en: URL: https://nutricionpersonalizada.wordpress.com/2011/04/26/epigenetica_diabetes_mellitus_tipo_2/. Último acceso: 23 de marzo de 2015.
11. Soriano-Tárraga C, Jiménez-Conde J, Giralt-Steinhauer E, Mola-Caminal M, Vivanco-Hidalgo RM, Ois A, et al. Asociación epigenética del gen TXNIP a diabetes mellitus tipo 2 y a hiperglicemia. Revista Genética Médica 2016;43:1-3.
Figura 1. Dinamismo del epigenoma con el paso del tiempo
.jpg)
Figura 2. Metilación del ácido desoxirribonucleico
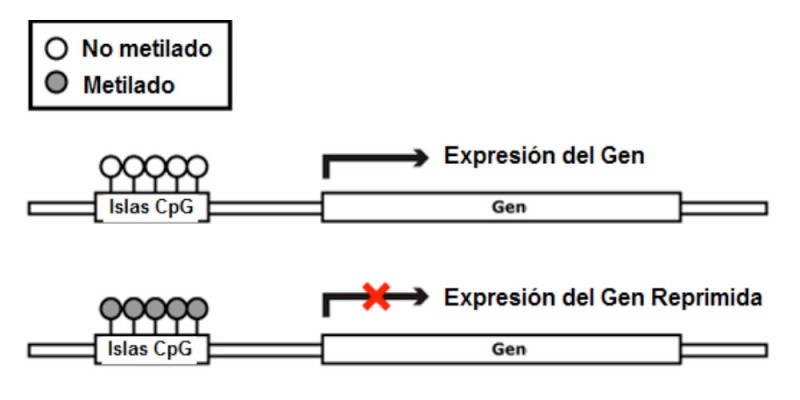
Figura 3. La unidad básica de la cromatina: el nucleosoma
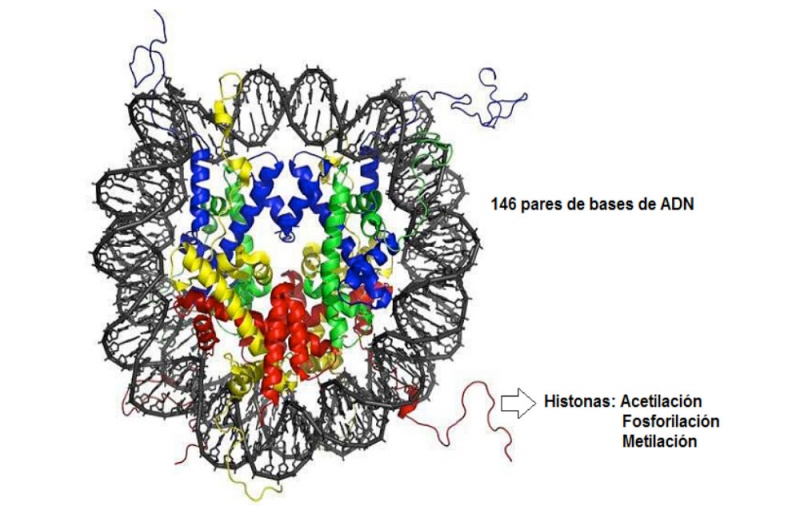
Figura 4. Modificación de histonas
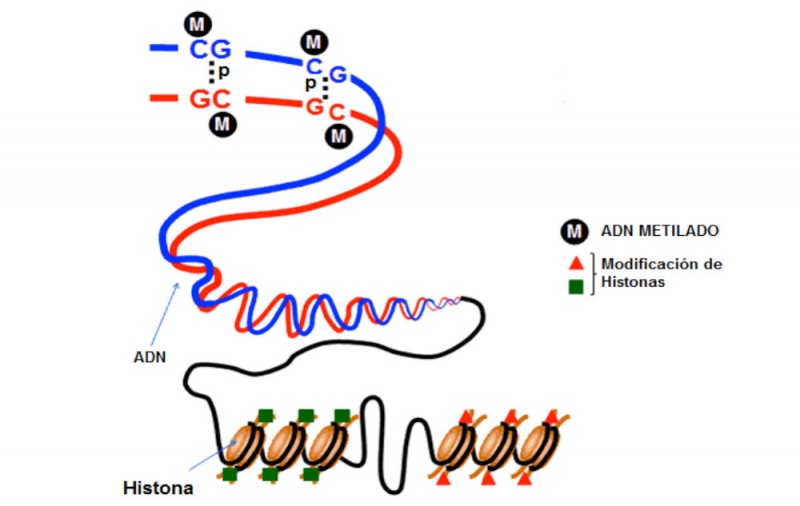
Figura 5. Interrupción de la traducción de proteínas por ARN no codificante